蛋白质接触图(Protein contact map,PCM)包含了重要的蛋白质空间几何约束信息,有助于解决诸多蛋白质结构相关的生物信息学问题,如蛋白质折叠[1, 2]、3D结构建模[3]和药物设计[4]等。使用传统的湿实验方法来测定蛋白质接触图非常耗时费力。因此,研发蛋白质接触图预测的自动化计算方法成为当下迫切的需求。近年来,用于蛋白质接触图预测的计算方法已经有了长足的发展[5, 6]。
在蛋白质中,当两个残基的Cβ原子(甘氨酸的情况为Cα)之间的空间距离小于一定的阈值(如8 Å),则认为这两个残基是相互接触的[6, 7]。一个蛋白质的接触图通常可以用一个对称矩阵来表示,矩阵中各元素的值为0或1,分别对应于非接触对或接触对。根据残基间的序列间距的不同,残基接触可以划分为短程、中程和长程3种类型之一。短程、中程和长程序列间距范围的标准分别为6~11,12~23和≥24,该标准已经广泛应用于包括CASP(Critical assessment of techniques for protein structure prediction)竞赛等相关领域[6, 7]。
在3种类型的残基接触中,中长程接触对于蛋白质的折叠起着至关重要的作用,因而引起了人们的广泛关注[8]。遗憾的是,虽然蛋白质接触预测取得了重大进展,但对于中长程接触的预测精度仍不尽如人意,远不能满足蛋白质三维结构建模等实际应用的需要[5]。在最近的CASP竞赛中(CASP 9和CASP 10),对于那些自由建模的蛋白质预测对象,即使是那些最先进的方法,长程接触预测的Top L/5的平均精度仍然低于13%[9, 10]。因此,研发能够进行高精度中长程接触图预测的新方法就有着迫切需求。
现阶段,接触图预测方法可大致分为两类:基于模板的方法和基于序列的方法[6]。对于一个待预测的蛋白质,基于模板的方法使用结构已知的蛋白质模板进行同源建模或线程化来预测蛋白质的接触图;基于序列的方法则是利用序列衍生特征来进行接触图的预测[11, 12]。基于模板的方法在很大程度上依赖于已知结构的模板的质量,当模板的质量欠缺时,往往不能得到令人满意的预测精度。因此,基于序列的方法受到越来越多的关注,特别是在海量蛋白质序列快速累积的后基因组时代。
基于序列的接触图预测方法又可以细分为两个子类,即基于协同进化的方法和基于机器学习的方法。基于协同进化的方法,比如PSICOV[13],GREMLIN[14],FreeContact[15]和CCMpred[16],均是基于接触残基共突变的假设,通过寻找多序列比对中的相关突变残基对来预测蛋白质残基接触;而基于机器学习的方法,比如BETACON[17],SVMcon[5],SVMSEQ[18],CMAPpro[19],PhyCMAP[20],Evfold[21]和NNcon[22],则是使用结构已知的蛋白质接触图作为训练数据,进而利用蛋白质序列特征训练预测模型来进行残基接触的预测工作。
基于协同进化的方法很大程度上依赖于从底层蛋白质数据库中获得的同源序列的数目和多样性。如果底层蛋白质数据库能够提供高质量的多序列比对(Multiple sequence alignments,MSA),则可以通过基于协同进化的方法实现对目标蛋白质序列进行可靠并且具有宽广覆盖域的接触图预测[13, 23, 24]。但是,如果目标序列获得的是质量较低的多序列比对,则不能得到令人满意的预测。基于机器学习的方法有较好的鲁棒性,不依赖于大量的多样化同源序列;但是,对于那些有大量同源序列的蛋白质,这些方法的预测精度往往要低于基于协同进化的方法。此外,因为训练样本里短程接触样本的数量远远大于长程接触样本,所以基于机器学习的方法预测出的接触图的覆盖域往往较窄[6, 23]。
如上所述,基于协同进化和基于机器学习的方法各有其优缺点。因此,通过集成学习将两类方法进行融合,有望提升蛋白质接触图预测的精度。近年来,已经出现了一些融合多个基于协同进化的和(或)基于机器学习的集成方法。其中,最具有代表性的集成预测方法为MetaPSICOV[25]和R2C[6]。最近,文献[26]提出了一个先进的集成方法NeBcon,该方法首先使用朴素贝叶斯分类器(Naïve Bayes classifier,NBC)融合8个现有的基于协同进化和基于机器学习的预测器,然后将NBC模型的输出与结构特征相结合,并在此基础上,应用一个简单的反向传播网络训练得到最终的接触图预测模型。
虽然NeBcon[26]在接触图预测方面取得了显著的进展,但是仍有进一步提升的空间。首先,NeBcon所融合的8个现有基础预测器之间可能存在冗余;其次,NeBcon使用NBC进行融合,NBC对每个基础预测器赋予相同的权重。而事实上,8个基础预测器的预测质量可能有所不同,通过学习自动获得权重较为合理。因此,使用加权朴素贝叶斯分类器(Weighted Naïve Bayes classifier, WNBC)[27-29]而不是NBC更符合实际情况;再次,可尝试其他先进的学习算法代替BP神经网络训练模型进一步提高预测性能。鉴于此,本文提出了NeBcon的增强版本,称为TargetPCM。与NeBcon相比,TargetPCM首先将8个基础预测器减为3个并采用加权朴素贝叶斯方法融合,而且使用粒子群算法(Particle swarm optimization,PSO)优化得到各分类器的权重系数[30-32]。然后,将WNBC的输出与基于序列的特征相结合形成新的特征向量。最后,应用极端随机树(Extremely randomized trees,ERT)训练得到最终的预测模型(TargetPCM)[33]。
1 实验材料与方法 1.1 基准数据集本文采用NeBcon[26]等使用的训练数据集Train_517。Train_517中包含517个蛋白质序列,并且任意两条序列之间的同源性低于25%。测试数据集分别采用NeBcon[26]使用的Test_98数据集及CASP11官网(http://predictioncenter.org/casp11/)中获得的测试数据集(下文中称为CASP11数据集)。Test_98数据集由98个两两序列同源性小于25%的非冗余蛋白质组成。CASP11数据集包括103个CASP11蛋白质,是在原始的105个CASP11的蛋白质基础上移除了2个残基数小于50的蛋白质(T0759-D1和T0820-D2)。103个CASP11蛋白质中,有48个被认为是难预测的。原因在于:对于这48个蛋白质中的任意一个,参与竞赛的前50%的预测服务器预测出的TM-score平均得分低于0.5[6]。
1.2 预测模型的结构框架TargetPCM使用基于粒子群优化算法的加权朴素贝叶斯方法(Weighted Naïve Bayes classifier based on particle swarm optimization,WNBC-PSO)融合3个互补的预测器(即基于机器学习的预测器(RFcon)、基于协同进化的预测器(PSICOV[13])和基于集成学习的预测器(MetaPSICOV[25])。特别地,RFcon是本文通过复现SVMcon[5]并将其中的支持向量机算法[34]替换成了随机森林算法[35]得到的。
图 1给出了接触图预测器TargetPCM的结构框图。对于1条蛋白质序列,首先使用3个基础预测器对其预测;接着通过WNBC-PSO融合3个基础预测器的输出从而得到基于预测器的特征,然后与从序列中提取的特征组合得到更有判别力的特征;在此基础上,应用ERT算法训练得到最终的预测模型来进行蛋白质接触图预测。
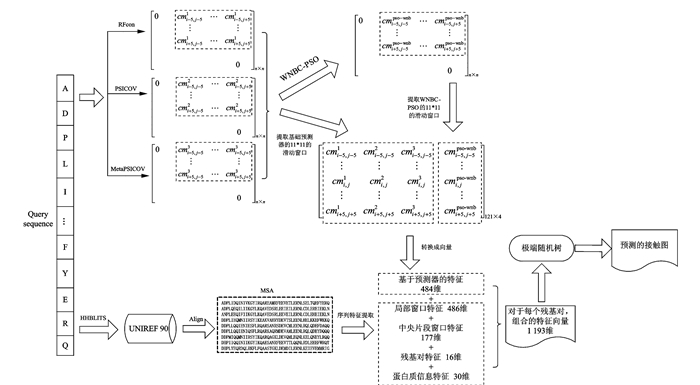
|
图 1 TargetPCM结构框架 Figure 1 The framework of TargetPCM |
1.3 用WNBC-PSO提取基于预测器的特征 1.3.1 加权朴素贝叶斯分类器
由于朴素贝叶斯分类器(NBC)的条件独立性假设在实际情况中很难得到满足,朴素贝叶斯分类器认为所有属性对决策的分类重要性程度是相同的,即权重均为1。而事实上,不同属性对分类的影响也是有差别的。
WNBC[27-29]是传统的NBC的一个扩展。相对于NBC,WNBC考虑各个基础分类器的重要性程度赋予其不同的权重。
假设N是基础接触图预测器的数目,对于一条蛋白质查询序列中的每个残基对,可以得到N个预测得分,用N维向量X=(x1, x2, …, xn, …, xN)T表示,其中xn是由第n个基础预测器预测得到的接触得分。假设C={C1, C2, …, Cm, …, CM}是M个类标签的集合。那么,残基对属于Cm类的后验概率公式为
| $ \begin{array}{l} P({C_m}|\mathit{\boldsymbol{X}}) = \frac{{P({C_m})P(\mathit{\boldsymbol{X}}|{C_m})}}{{P\left( \mathit{\boldsymbol{X}} \right)}} = \\ \;\;\;\;\;\;\;\;\;\frac{{P({C_m})\prod\limits_{n = 1}^N {P{{({x_n}|{C_m})}^{{w_n}}}} }}{{P\left( \mathit{\boldsymbol{X}} \right)}} \end{array} $ | (1) |
式中:P(Cm)是Cm类的先验概率;P(X|Cm)是Cm类中X的条件概率;P(X)是证据因子,并且wn是第n个基础预测器的权重系数。
在本文中,Cm∈{0, 1},其中1和0分别代表残基对是接触的或是不接触的。因此,残基对属于接触类的后验概率表示为
| $ \begin{array}{l} P\left( {1|\mathit{\boldsymbol{X}}} \right) = \frac{{P\left( 1 \right)P\left( {\mathit{\boldsymbol{X}}|1} \right)}}{{P\left( \mathit{\boldsymbol{X}} \right)}} = \\ \frac{{P\left( 1 \right)\prod\limits_{n = 1}^N {P{{({x_n}|1)}^{{w_n}}}} }}{{P\left( 1 \right)\prod\limits_{n = 1}^N {P{{({x_n}|1)}^{{w_n}}}} + P\left( 0 \right)\prod\limits_{n = 1}^N {P{{({x_n}|0)}^{{w_n}}}} }} \end{array} $ | (2) |
基于训练数据集Train_517,可以使用NeBcon[26]中描述的方法计算得到在式(2)中的4个参数,即P(0),P(1),P(xn|1)和P(xn|0)。另外,针对短程、中程和长程接触,需要分别计算这4个参数来加强模型的特异性[26]。
1.3.2 使用PSO算法优化WNBC的权重本文使用PSO优化WNBC中每个基础分类器的权重[30-32, 36, 37]。实验中,1组基础接触图预测器的权重被编码成1个候选粒子,并且将WNBC在训练集Train_517上的Top L/5的平均精度作为适应度函数。WNBC-PSO的过程简述如下:
步骤1 设定粒子群的规模和最大迭代次数,并且随机初始化粒子群中每个粒子的位置。
步骤2 标准化每个粒子的位置作为基础分类器的权重,使用WNBC融合各个基础分类器并对数据集Train_517分类预测,计算得到每个粒子的适应度,来更新个体极值和全局极值,从而调整粒子群中每个粒子的位置和速度。
步骤3 重复上述过程直到达到最大迭代次数,迭代结束,找到最优粒子。
1.3.3 基于预测器的特征提取对于1条蛋白质查询序列中的每个残基对(i, j),都可以得到1个4维向量,其中的4个元素分别是3个基础预测器(即RFcon,PSICOV[13]和MetaPSICOV[25])预测的接触得分和WNBC-PSO融合的后验概率。另外,为了提高特征的鲁棒性,设置了分别以残基i和残基j为中心的11个残基的滑动窗口[5, 26]。这样,对于每个残基对(i, j),通过组合两个窗口中的所有残基对的四维向量可以得到1个484维(484=11×11×4)的基于预测器的特征向量。
1.4 基于序列的特征提取除了基于预测器的特征,本文对每个残基对(i, j)提取基于序列的特征。实验中,采用SVMcon[5]中的方法提取基于序列的特征,其中包括486维的局部窗口特征,177维的中央片段窗口特征,16维的残基对特征以及30维的蛋白质信息特征,基于序列的特征的详细提取方法参见文献[5]。
1.5 训练ERT预测模型本文组合使用了基于预测器的特征和基于序列的特征训练ERT预测模型。首先,将每个残基对的序列特征和预测器特征组合形成最终的1 193(=486+177+16+30+484)维的特征向量。然后,基于这样鲁棒的特征表示,进一步应用极端随机树(ERT)[33]在训练集Train_517上训练预测模型。对于短程和中程接触类型,所有接触的残基对的特征向量组成正训练样本集,同时所有非接触的残基对的特征向量组成负训练样本集。对于长程接触,由于训练样本集过大,因此本文构建了一个100万的长程训练数据集,其中包括所有的长程接触样本以及部分随机采样的长程非接触样本。为了加强训练模型的特异性,对于短程、中程和长程接触,需要分别训练ERT模型(由scikit-learn[38]实现)。
2 结果与讨论本文通过与其他主流的接触图预测器的性能进行比较分析,系统地评估TargetPCM的性能。评价过程中,采用得分最高的前K个预测的精确度Accuracy作为评价指标,这也是本领域广泛应用的评价指标。
对于1个包括L个残基的蛋白质,可得到(L+1-6)×(L-6)/2个残基对,其中6是在实验中所使用的序列间距的最小值。一个训练好的预测器首先预测每个残基对接触的概率得分然后对所有预测的得分进行降序排序。之后,使用Accuracy=Ncorr(K)/K计算得分最高的前K个预测的Accuracy来评估预测器的性能,其中Ncorr(K)是得分最高的前K个预测中正确预测的残基对数目。另外,由于真实接触的总数量与蛋白质长度近似成线性关系[39],因此L,L/2,L/5和L/10是K的几个常用值。
2.1 WNBC-PSO组合预测器性能分析首先研究通过WNBC-PSO组合多个预测器的预测性能。在训练集Train_517上,分别使用朴素贝叶斯分类器(NBC)和基于粒子群算法的加权朴素贝叶斯分类器(WNBC-PSO)组合3个基础预测器(PSICOV[13],RFcon和MetaPSICOV[25])。实验中,粒子群的规模和最大迭代次数分别设为10和100,并针对短程、中程和长程接触分别单独优化WNBC的权重。另外,本文尝试增大粒子群规模或是增加最大迭代次数,发现WNBC的权重并无显著变化,反而增加了实验的额外开销,表明这两个实验参数的设置对于优化WNBC的权重是有效的。
在包括98个蛋白质序列的Test_98和103个蛋白质序列的CASP11上,对两个训练好的集成预测器(NBC和WNBC-PSO)进行测试,表 1汇总了两个集成预测器和3个基础预测器的Top L/5的预测精度。PSICOV和MetaPSICOV的结果是将测试序列输入到从各自的网站上下载下来的训练模型中得到的;RFcon的结果是将测试序列输入到在SVMcon的训练数据集上训练的模型中得到的;而NBC和WNBC-PSO的结果是将测试序列输入到在Train_517上训练的相应模型中得到的。
| 表 1 在Test_98和CASP11测试集上3个基础预测器以及两个集成预测器的Top L/5的预测精度 Table 1 Accuracy of Top L/5 contact predictions by three base predictors and two combined predictors on the 98 targets in Test_98 and the 103 CASP11 targets |
从表 1中,可以得到如下两点结论:
(1) 通过集成多个互补的预测器,接触图预测的精度得到进一步提高。和3个独立的基础预测器(RFcon,PSICOV和MetaPSICOV)比较,对于短程、中程和长程接触两个集成预测器(NBC和WNBC-PSO)都取得了显著的性能提升。分析取得性能提升的可能原因是集成预测器充分利用了独立的基础预测器所提供的互补信息,从而提升了整体的预测精度。
(2) 对于短程、中程和长程接触,NBC算法的改进版WNBC-PSO均取得了最优的预测性能以及最高的精度,WNBC-PSO在Test_98上的精度分别为66.84%,64.71%和63.71%,在CASP11上的精度为60.02%,60.19%和56.59%。WNBC-PSO取得比NBC更优越性能的原因是WNBC-PSO使用PSO来学习3个基础预测器的权重,合理地赋予基础预测器恰当的权重,有助于区分基础预测器的重要性程度,从而加强整体的预测性能。而NBC算法赋予给不同的基础分类器的权重是相等的,即认为每个基础分类器对最终分类决策的影响是一致的,这种方式无法区分效果较差和效果较好的预测器对分类的重要性程度,从而降低整体的预测效果。
2.2 TargetPCM预测模型性能分析为了更进一步地提高接触图预测的性能,本文组合基于预测器的特征及基于序列的特征训练ERT预测模型,称为TargetPCM。实验中两个重要参数,即生成树的数目(nTree)和在每次分裂时随机抽取为候选的维数(mTry)都是分别设置为500和60。表 2在测试集Test_98和CASP11上比较了WNBC-PSO和TargetPCM之间的Top L/5的预测精度。
| 表 2 在Test_98和CASP11测试集上,WNBC-PSO和TargetPCM的Top L/5的预测精度比较 Table 2 Comparison of accuracy of Top L/5 predictions for short-, medium-, and long-range contacts between WNBC-PSO and TargetPCM on Test_98 and CASP11 |
如表 2所示,在Test_98和CASP11测试集上,TargetPCM的预测精度得到显著的提升。在Test_98上,对于短程、中程和长程接触TargetPCM取得的精度分别是70.43%,66.65%和66.11%,比WNBC-PSO的精度提升了3.59%,1.94%和2.40%。而且同样可以在CASP11上观察到相似的精度提升。分析TargetPCM优于WNBC-PSO的原因如下:
由于TargetPCM结合了从WNBC-PSO得到的基于预测器的特征(后验概率得分)以及每个残基对的基于序列的特征。这个鲁棒的特征表示方法加强了用于接触图预测的残基对的特征判别能力,并且结合ERT模型强大的分类能力,因此TargetPCM可以取得更卓越的接触图预测性能。
2.3 与主流接触图预测器比较 2.3.1 在Test_98测试集上比较为了进一步评估本文提出的TargetPCM的预测性能,本文将它与主流的接触图预测器比较,包括基于机器学习的预测器(RFcon,BETAcon[17],SVMcon[5]和SVMSEQ[18]),基于协同进化的预测器(CCMpred[16],PSICOV[13]和FreeContact[15])以及基于集成学习的预测器(MetaPSICOV[25],NeBcon[26]和TargetPCM),比较结果如表 3所示。SVMcon,SVMSEQ,BETAcon,FreeContact,PSICOV,CCMpred和MetaPSICOV的结果是将测试序列输入到从各自网站下载的训练模型中得到的;NeBcon的结果是摘自文献[26];RFcon的结果是将测试序列输入到在SVMcon的训练数据集上训练的模型中得到的;TargetPCM的结果是将测试序列输入到在Train_517上训练的相应模型中得到的。
| 表 3 在Test_98测试集上,TargetPCM和其他主流的预测器的Top L/5接触预测精度的比较 Table 3 Comparison of accuracy of Top L/5 contact predictions between TargetPCM and other existing predictors on Test_98 |
从表 3中可知:
(1) 对于短程和中程的接触,基于机器学习的接触预测器比基于协同进化的预测器性能更好。
对于短程接触,4个基于机器学习的预测器的平均精度是51.86%,比3个基于协同进化的预测器的平均精度(即30.11%)高21.75%。
对于中程接触,4个基于机器学习的预测器的平均精度比3个基于协同进化的预测器的平均精度高8.21%。
(2) 对于长程接触,基于机器学习的预测器的预测性能不如基于协同进化的预测器,4个基于机器学习的预测器的平均精度仅仅只有27.84%,明显低于3个基于协同进化的预测器的平均精度42.29%。
(3) 3个集成预测器相比起基于机器学习和基于协同进化的预测器具有压倒性优势。对于短程、中程和长程接触,3个集成预测器的平均精度分别达到了66.39%,62.25%和63.29%。这些结果显著表明了集成多个独立的和互补的预测器是进一步提高接触预测精度的一条有效途径。
(4) 对于短程、中程和长程接触,TargetPCM比NeBcon的预测精度均有显著提高。虽然TargetPCM和NeBcon[26]有着相似的预测结构,但是在预测精度上,对于短程、中程和长程接触,TargetPCM比NeBcon的预测精度分别提高了8.2%(=(70.43-65.10)/65.10),16.1%(=(66.65-57.40)/57.40)和5.3%(=(66.11-62.80)/62.80),TargetPCM优于NeBcon的原因包括如下5个方面:
(1) TargetPCM融合了3个互补的具有代表性的基础预测器来生成基于预测器的特征。NeBcon融合了8个基础预测器,其中部分基础预测器的效果较差以及互相之间可能存在冗余,从而导致整体的预测性能降低。
(2) TargetPCM采用PSO算法优化各分类器的权重。训练过程中,TargetPCM为不同的分类器分配不同的基础权重,而NeBcon使用NBC组合8个基础预测器,并给每个预测器赋予相同的权重。显然,不同的基础预测器具有不同的预测质量,若分配相同的权重,会使得预测效果较差的预测器和较优的预测器对融合的结果具有相同的地位,从而降低整体的预测性能,因此分配适当的权重更为合理。TargetPCM仔细考虑各个基础预测器的重要性差别,所以使用更合理的WNBC融合基础预测器,并使用PSO算法优化每个基础预测器的权重,从而提高整体的预测性能。
(3) TargetPCM包括的基于序列的特征达到了709维,虽然NeBcon所提取的基于序列的特征也有596维,但仍不如TargetPCM。因此更广泛的序列信息使得TargetPCM在训练时能学习到更有效的规律,从而提高模型的分类性能。
(4) TargetPCM含有的基于预测器的特征不仅仅包括加权朴素贝叶斯分类器的后验概率,而且还包括3个基础分类器(RFcon,PSICOV和MetaPSICOV)的接触得分,而NeBcon仅仅只有朴素贝叶斯分类器的后验概率。因此TargetPCM提取的基于预测器的特征达到了484维,而NeBcon只有121维,对预测器信息的有效利用使得TargetPCM具有更强大的分类判别能力。
(5) TargetPCM基于极端随机树算法训练最终的预测模型。极端随机树算法是1种分类效果出色的集成学习算法,相对于NeBcon中使用的反向传播算法,更适合处理高维特征的样本数据。因此分类效果出色,而且能够实现并行化计算,这也是TargetPCM取得更优异性能的一个重要原因。
2.3.2 在CASP11测试集上比较本文在CASP11测试集上与其他主流的接触图预测器进行比较,验证TargetPCM的优越性。在预测器的选择上,RFcon和PSICOV[13]分别是基于机器学习和基于协同进化中具有代表性的两个预测器,R2C[6]和MetaPSICOV[25]是效果比较出类拔萃的集成预测器。对于CASP11中的103个蛋白质,NeBcon[26]仅仅提供了带有自由建模域的33个蛋白质的预测结果,因此不包括它作比较。
表 4汇总了在CASP11的103个蛋白质上,本文的TargetPCM和其他现有的预测器的Top L/5预测精度的比较,表 5汇总了在CASP11的48个难预测的蛋白质上与其他预测器的Top L/5预测精度的比较。表 4和表 5中的结果是按照如下方式获得的:PSICOV和MetaPSICOV的结果是将测试序列输入到从各自的网站上下载下来的训练模型中得到的;RFcon的结果是将测试序列输入到在SVMcon的训练数据集上训练的模型中得到的;NBC和WNBC-PSO的结果是将测试序列输入到在Train_517上训练的相应模型中得到的;R2C的结果是摘自文献[6];TargetPCM的结果是将测试序列输入到在Train_517上训练的相应模型中得到的。
| 表 4 在CASP11的103个蛋白质上,TargetPCM和其他现有预测器的Top L/5接触预测精度的比较 Table 4 Comparison of accuracy of Top L/5 contact predictions between TargetPCM and other existing predictors on the 103 targets in CASP11 |
| 表 5 在CASP11的48个难预测的蛋白质上,TargetPCM和其他现有预测器的Top L/5接触预测精度的比较 Table 5 Comparison of accuracy of Top L/5 contact predictions between TargetPCM and other existing predictors on the 48 hard targets in CASP11 |
从表 4和表 5中可知,本文提出的TargetPCM不仅仅远远优于PSICOV[13]和RFcon,而且还领先于MetaPSICOV[25]和R2C[6]。在CASP11的103个蛋白质上,对于短程、中程和长程接触TargetPCM分别获得了63.06%,63.91%和58.81%的精度,即TargetPCM的Top L/5精度比最好的基于协同进化的集成预测器(MetaPSICOV[25])分别提高了7.4%(=(63.06-58.70)/58.70),9.1%(=(63.91-58.59)/58.59)和7.5%(=(58.81-54.73)/54.73)。
在CASP11的48个难预测的蛋白质上,对于短程、中程和长程接触本文提出的TargetPCM依然分别获得57.78%,56.40%和38.92%的最卓越的预测效果。因此表 4和表 5中的结果进一步表明了本文提出的TargetPCM的有效性。
2.4 案例研究本文采用在CASP11测试集中的蛋白质(T0769-D1)来直观地表明本文所提出的TargetPCM相比起MetaPSICOV[25]具有更优越的预测性能。T0769-D1包括97个残基(即L=97)并且拥有143个真实的长程接触。图 2根据长程接触,对于蛋白质T0769-D1分别绘制了MetaPSICOV和TargetPCM的真实的和预测的对比图。上三角的十字标记代表在Top L预测中的真实的长程接触,然而在下三角的十字标记代表在Top L预测中的预测的长程接触。
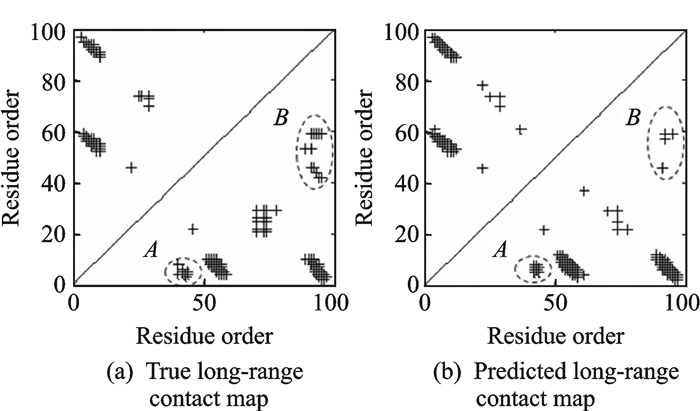
|
图 2 MetaPSICOV和TargetPCM对于蛋白质T0769-D1的真实的和预测的长程接触图 Figure 2 True versus predicted long-range contact maps of MetaPSICOV and TargetPCM for protein T0769-D1 |
在Top L的预测中,MetaPSICOV预测出了44个真实的长程接触,而TargetPCM正确预测出了56个真实的长程接触。在图 2中,TargetPCM比MetaPSICOV的预测性能更加优异。比如,在MetaPSICOV和TargetPCM的预测中存在两个假正样本数据簇(即图 2的两个子图中的簇A和簇B)。在两个簇中,MetaPSICOV预测的长程接触的数量明显多于TargetPCM,这表明在MetaPSICOV的长程接触预测中许多负样本被预测为正样本。
3 结束语本文提出了一种基于WNBC-PSO和ERT的用于蛋白质残基接触图预测的集成预测器(TargetPCM)。TargetPCM使用WNBC-PSO组合了RFcon,PSICOV[13]和MetaPSICOV[25],它们分别是基于机器学习、基于协同进化和基于集成的预测器,组合这些不同类型的预测器能够充分利用它们提供的互补信息。为了进一步提高预测性能,本文将WNBC-PSO的输出(即基于预测器的特征)和蛋白质基于序列的特征组合来形成更有判别力的特征,基于这个鲁棒的特征表示方法,训练得到最终的ERT预测模型(TargetPCM)。
为了验证TargetPCM的预测性能,本文将TargetPCM在Test_98和CASP11这两个独立的数据集上测试,发现对于短程、中程和长程接触,TargetPCM在Test_98上的Top L/5的预测精度比最好的集成预测器(NeBcon)分别提高了8.2%,16.1%和5.3%。在CASP11数据集上,对于短程、中程和长程接触,TargetPCM的Top L/5的预测精度比最好的基于协同进化的集成预测器(MetaPSICOV)分别提高了7.4%,9.1%和7.5%,这些比较表明了本文提出的TargetPCM对于蛋白质接触图预测的有效性。
尽管TargetPCM取得了良好的预测性能,但仍有进一步改进的空间。将来的工作将研究更有效的特征提取方法和机器学习方法(比如深度学习[40])来进一步提高蛋白质接触图的预测性能。
| [1] |
CHENG J, BALDI P.
A machine learning information retrieval approach to protein fold recognition[J]. Bioinformatics, 2006, 22(12): 1456–1463.
DOI:10.1093/bioinformatics/btl102
|
| [2] |
OLMEA O, ROST B, VALENCIA A.
Effective use of sequence correlation and conservation in fold recognition[J]. Journal of Molecular Biology, 1999, 293(5): 1221–1239.
DOI:10.1006/jmbi.1999.3208
|
| [3] |
BONNEAU R, RUCZINSKI I, TSAI J, et al.
Contact order and ab initio protein structure prediction[J]. Protein Science a Publication of the Protein Society, 2002, 11(8): 1937–1944.
DOI:10.1110/ps.3790102
|
| [4] |
KLIGER Y, LEVY O, OREN A, et al.
Peptides modulating conformational changes in secreted chaperones:From in silico design to preclinical proof of concept[J]. Proceedings of the National Academy of Sciences, 2009, 106(33): 13797–13801.
DOI:10.1073/pnas.0906514106
|
| [5] |
PIERRE B, CHENG J.
Improved residue contact prediction using support vector machines and a large feature set[J]. BMC Bioinformatics, 2007, 8(1): 1–9.
DOI:10.1186/1471-2105-8-1
|
| [6] |
YANG J, JIN Q Y, ZHANG B, et al.
R2C:Improving ab initio residue contact map prediction using dynamic fusion strategy and Gaussian noise filter[J]. Bioinformatics, 2016, 32(16): 2435–2443.
DOI:10.1093/bioinformatics/btw181
|
| [7] |
LI Y, FANG Y, FANG J.
Predicting residue-residue contacts using random forest models[J]. Bioinformatics, 2011, 27(24): 3379–3384.
DOI:10.1093/bioinformatics/btr579
|
| [8] |
GROMIHA M M.
Influence of long-range contacts and surrounding residues on the transition state structures of proteins[J]. Analytical Biochemistry, 2011, 408(1): 32–36.
DOI:10.1016/j.ab.2010.08.029
|
| [9] |
MONASTYRSKYY B, D'ANDREA D, FIDELIS K, et al.
Evaluation of residue-residue contact prediction in CASP10[J]. Proteins-Structure Function & Bioinformatics, 2014, 82(S2): 138–153.
|
| [10] |
MONASTYRSKYY B, D'ANDREA D, FIDELIS K, et al.
New encouraging developments in contact prediction:Assessment of the CASP11 results[J]. Proteins-Structure Function & Bioinformatics, 2015, 84(S1): 131–144.
|
| [11] |
SKOLNICK J, KIHARA D, ZHANG Y.
Development and large scale benchmark testing of the PROSPECTOR_3 threading algorithm[J]. Proteins-Structure Function & Bioinformatics, 2004, 56(3): 502–518.
|
| [12] |
MISURA K M, CHIVIAN D, ROHL C A, et al.
Physically realistic homology models built with rosetta can be more accurate than their templates[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(14): 5361–5366.
DOI:10.1073/pnas.0509355103
|
| [13] |
JONES D T, BUCHAN D W, COZZETTO D, et al.
PSICOV:Precise structural contact prediction using sparse inverse covariance estimation on large multiple sequence alignments[J]. Bioinformatics, 2012, 28(2): 184–190.
DOI:10.1093/bioinformatics/btr638
|
| [14] |
KAMISETTY H, OVCHINNIKOV S, BAKER D.
Assessing the utility of coevolution-based residue-residue contact predictions in a sequence- and structure-rich era[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(39): 15674–15679.
DOI:10.1073/pnas.1314045110
|
| [15] |
KAJ N L, HOPF T A, KALAS M, et al.
FreeContact:Fast and free software for protein contact prediction from residue co-evolution[J]. BMC Bioinformatics, 2014, 15(1): 85.
DOI:10.1186/1471-2105-15-85
|
| [16] |
SEEMAYER S, GRUBER M, S DING J.
CCMpred——fast and precise prediction of protein residue-residue contacts from correlated mutations[J]. Bioinformatics, 2014, 30(21): 3128–3130.
DOI:10.1093/bioinformatics/btu500
|
| [17] |
CHENG J, BALDI P.
Three-stage prediction of protein β-sheets by neural networks, alignments and graph algorithms[J]. Bioinformatics, 2005, 21(S): 75–84.
|
| [18] |
WU S, ZHANG Y.
A comprehensive assessment of sequence-based and template-based methods for protein contact prediction[J]. Bioinformatics, 2008, 24(7): 924–931.
DOI:10.1093/bioinformatics/btn069
|
| [19] |
DI L P, NAGATA K, BALDI P.
Deep architectures for protein contact map prediction[J]. Bioinformatics, 2012, 28(19): 2449–2457.
DOI:10.1093/bioinformatics/bts475
|
| [20] |
WANG Z, XU J.
Predicting protein contact map using evolutionary and physical constraints by integer programming[J]. Bioinformatics, 2013, 29(13): 266–273.
DOI:10.1093/bioinformatics/btt211
|
| [21] |
MARKS D S, COLWELL L J, SHERIDAN R, et al.
Protein 3D structure computed from evolutionary sequence variation[J]. PLOS One, 2011, 6(12): e28766.
DOI:10.1371/journal.pone.0028766
|
| [22] |
TEGGE A N, WANG Z, EICKHOLT J, et al.
NNcon:Improved protein contact map prediction using 2D-recursive neural networks[J]. Nucleic Acids Research, 2009, 37(Web Server issue): 515–518.
|
| [23] |
YANG J, JANG R, ZHANG Y, et al.
High-accuracy prediction of transmembrane inter-helix contacts and application to GPCR 3D structure modeling[J]. Bioinformatics, 2013, 29(20): 2579–2587.
DOI:10.1093/bioinformatics/btt440
|
| [24] |
SKWARK M J, RAIMONDI D, MICHEL M, et al.
Improved contact predictions using the recognition of protein like contact patterns[J]. Plos Computational Biology, 2014, 10(11): e1003889.
DOI:10.1371/journal.pcbi.1003889
|
| [25] |
JONES D T, SINGH T, KOSCIOLEK T, et al.
MetaPSICOV:Combining coevolution methods for accurate prediction of contacts and long range hydrogen bonding in proteins[J]. Bioinformatics, 2015, 31(7): 999–1006.
DOI:10.1093/bioinformatics/btu791
|
| [26] |
HE B, MORTUZA S M, WANG Y, et al.
NeBcon:Protein contact map prediction using neural network training coupled with naïve Bayes classifiers[J]. Bioinformatics, 2017, 33(15): 2296–2306.
DOI:10.1093/bioinformatics/btx164
|
| [27] |
WEBB G I, PAZZANI M J. Adjusted probability naive Bayesian induction[C]//Australian Joint Conference on Artificial Intelligence.[S.l.]: [s.n.], 1998: 285-295. |
| [28] |
ZHANG H, SHENG S. Learning weighted naive Bayes with accurate ranking[C]//IEEE International Conference on Data Mining.[S.l.]: IEEE, 2004: 567-570. |
| [29] |
邓维斌, 王国胤, 洪智勇.
基于粗糙集的加权朴素贝叶斯邮件过滤方法[J]. 计算机科学, 2011, 38(2): 218–221.
DOI:10.3969/j.issn.1002-137X.2011.02.051 DENG Weibin, WANG Guoyin, HONG Zhiyong. Weighted Naïve Bayes spam filtering method based on rough set[J]. Computer Science, 2011, 38(2): 218–221. DOI:10.3969/j.issn.1002-137X.2011.02.051 |
| [30] |
EBERHART R C.
A modified particle swarm optimizer[M]. Berlin, Heidelberg: Springer, 1998: 69-73.
|
| [31] |
KENNEDY J, EBERHART R. Particle swarm optimization[C]//IEEE International Conference on Neural Networks, 1995 Proceedings. Perth, WA, Australia: IEEE, 2002: 1942-1948. |
| [32] |
EBERHART R, KENNEDY J. A new optimizer using particle swarm theory[C]//International Symposium on MICRO Machine and Human Science. Nagoya, Japan: IEEE, 2002: 39-43. |
| [33] |
GEURTS P, ERNST D, WEHENKEL L.
Extremely randomized trees[J]. Machine Learning, 2006, 63(1): 3–42.
DOI:10.1007/s10994-006-6226-1
|
| [34] |
刘万里, 刘三阳, 王金艳.
不平衡支持向量机的调整方法[J]. 计算机科学, 2009, 36(3): 148–149.
DOI:10.3969/j.issn.1002-137X.2009.03.040 LIU Wanli, LIU Sanyang, WANG Jinyan. Adjusting method for imbalanced support vector machines[J]. Computer Science, 2009, 36(3): 148–149. DOI:10.3969/j.issn.1002-137X.2009.03.040 |
| [35] |
张洪强, 刘光远, 赖祥伟.
随机森林算法在肌电的重要特征选择中的应用[J]. 计算机科学, 2013, 40(1): 200–202.
DOI:10.3969/j.issn.1002-137X.2013.01.046 ZHANG Hongqiang, LIU Guangyuan, LAI Xiangwei. Application of random forest algorithm in important feature selection from EMG signal[J]. Computer Science, 2013, 40(1): 200–202. DOI:10.3969/j.issn.1002-137X.2013.01.046 |
| [36] |
高尚, 杨静宇.
一种新的基于粒子群算法的聚类方法[J]. 南京航空航天大学学报, 2006, 38(S1): 62–65.
GAO Shang, YANG Jingyu. New clustering method based on particle swarm algorithm[J]. Journal of Nanjing University of Aeronautics & Astronautics, 2006, 38(S1): 62–65. |
| [37] |
白俊杰, 王宁生, 唐敦兵.
一种求解多目标柔性作业车间调度的改进粒子群算法[J]. 南京航空航天大学学报, 2010, 42(4): 447–453.
DOI:10.3969/j.issn.1005-2615.2010.04.010 BAI Junjie, WANG Ningsheng, TANG Dunbing. Improved PSO algorithm for multi-objective optimization flexible job shop scheduling problems[J]. Journal of Nanjing University of Aeronautics & Astronautics, 2010, 42(4): 447–453. DOI:10.3969/j.issn.1005-2615.2010.04.010 |
| [38] |
PEDREGOSA F, GRAMFORT A, MICHEL V, et al.
Scikit-learn:Machine learning in python[J]. Journal of Machine Learning Research, 2012, 12(10): 2825–2830.
|
| [39] |
LUND O, FRIMAND K, GORODKIN J, et al.
Protein distance constraints predicted by neural networks and probability density functions[J]. Protein Engineering, 1997, 10(11): 1241–1248.
DOI:10.1093/protein/10.11.1241
|
| [40] |
LECUN Y, BENGIO Y, HINTON G.
Deep learning[J]. Nature, 2015, 521(7553): 436–444.
DOI:10.1038/nature14539
|
 2018, Vol. 50
2018, Vol. 50


